生信论文的套路
ONCOMINE从全景、亚型两个维度做表达差异分析;
临床标本从蛋白水平确认(或HPA数据库),很重要;
Kaplan-Meier Plotter从临床意义的角度阐明其重要性;
cBio-portal数据库做基因组学的分析(机制一);
STRING互作和GO/KEGG分析探讨可能的信号通路(机制二);
TISIDB/TIMER分析肿瘤免疫特征(机制三)。
我们前面说过,仅仅靠ONCOMINE数据库的数据,还很难发表论文。而且,ONCOMINE数据库的数据,是芯片或测序的结果,是从转录水平进行的检测。而差异表达,最好是在蛋白水平,因此,对于ONCOMINE数据库分析得到的结果,最好进行蛋白水平的验证。
检测蛋白表达的方法很多,但是鉴于生信分析的特殊性,有条件去做WB和免疫组化当然是最好的。如果没有条件,用人类蛋白组图谱数据库(The human preotein atlas, HPA,网址 https://www.proteinatlas.org/)是最方便的。无需注册,直接使用。
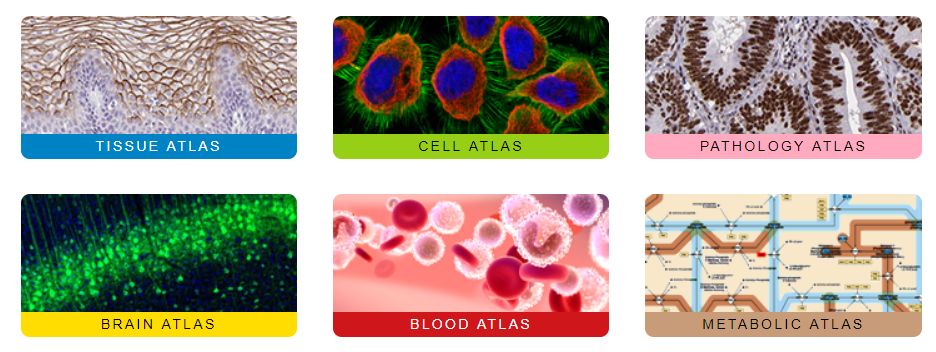
HPA基本上提供了人类可获得的所有蛋白(约26000种)的在人体组织、器官的表达和分布。数据库中,研究人员使用高度特异性的抗体(一般是多种抗体),用免疫组化(IHC)检测每种蛋白在64个细胞系、48种人类正常组织和20种肿瘤组织中的表达情况。
数据库从TISSUE ALTAS,CELL ALTAS和PATHOLOGY ALTAS角度分别展示蛋白在正常组织、细胞和肿瘤组织中的表达情况,还展示蛋白在脑组织(BRAIN ALTAS)、血液(BLOOD ALTAS)和代谢(METABLIC ATLAS)中的表达和可能的作用。功能很强大。
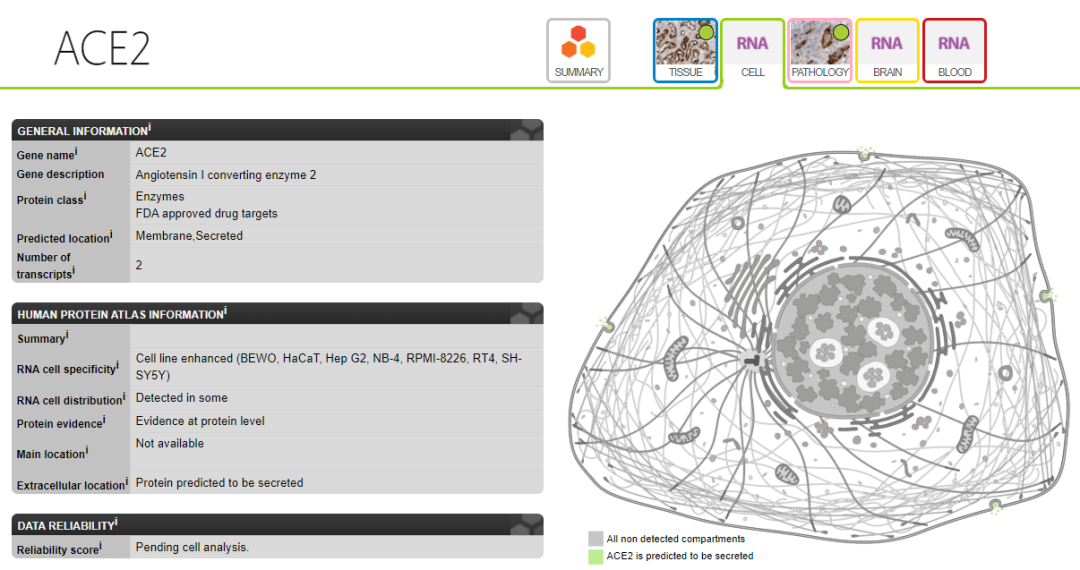
比如,我们以新冠病毒的受体ACE2为例,简要说明HPA的用法和数据呈现。输入ACE2,搜索。
在组织的表达,出现如下界面,RNA表达和蛋白表达的数据都有。
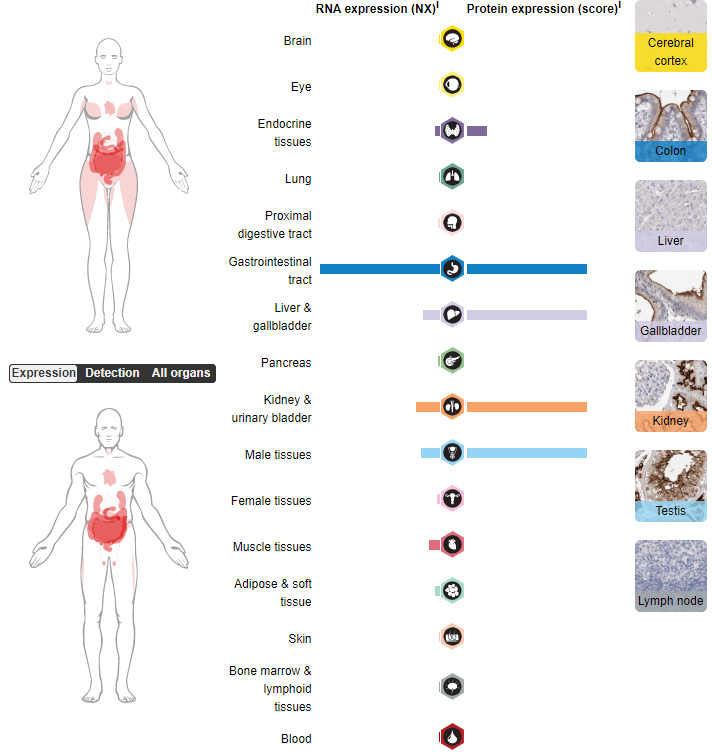
蛋白表达水平。
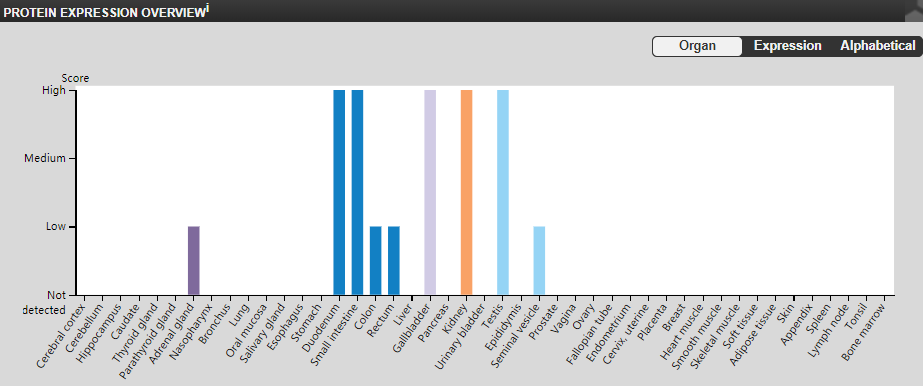
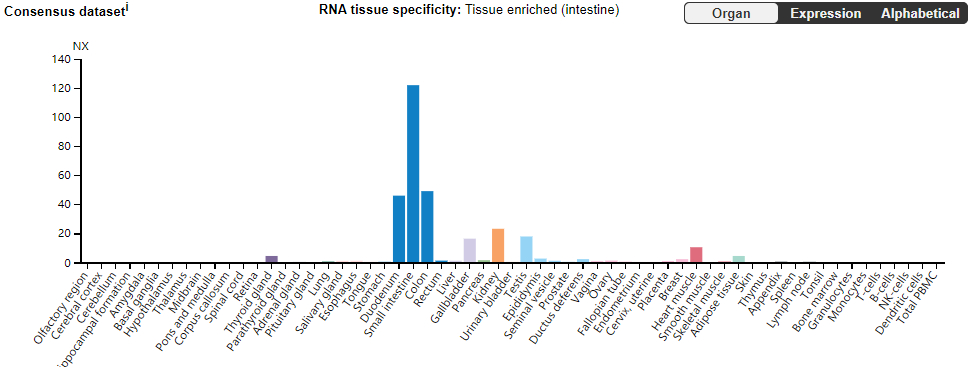
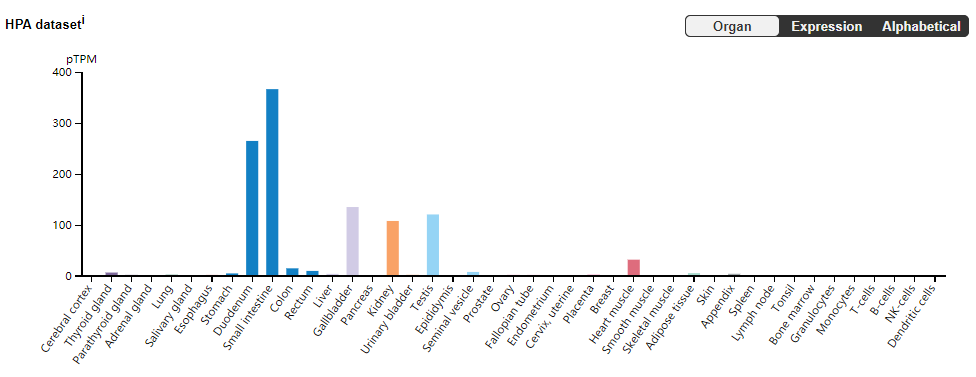
转录水平,从四个数据的结果来展示ACE2在人体组织的表达情况。
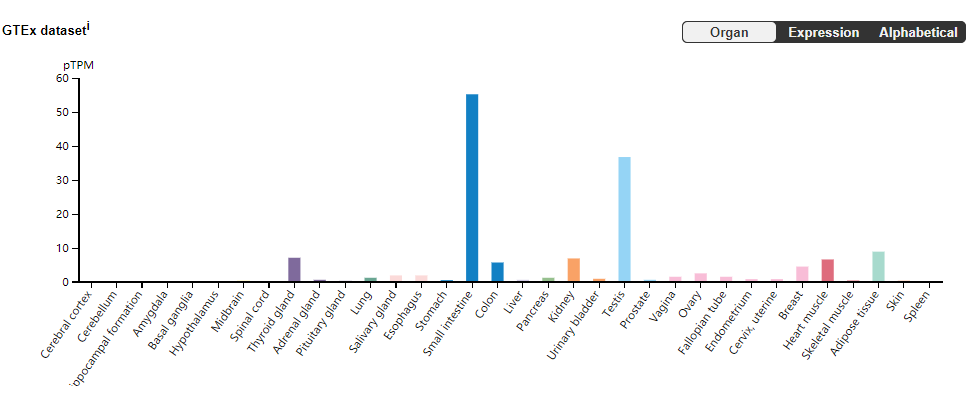
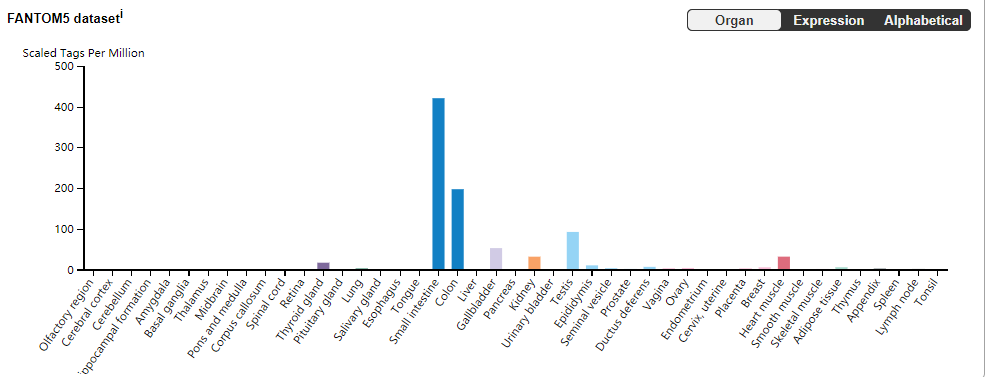
代谢功能的简介和总结

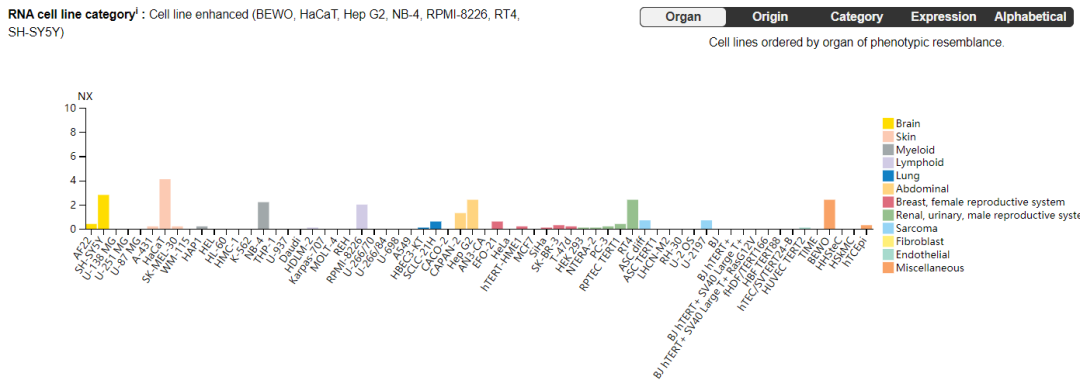
在细胞系中的表达如下。
在肿瘤组织的表达情况信息更多。

预后价值
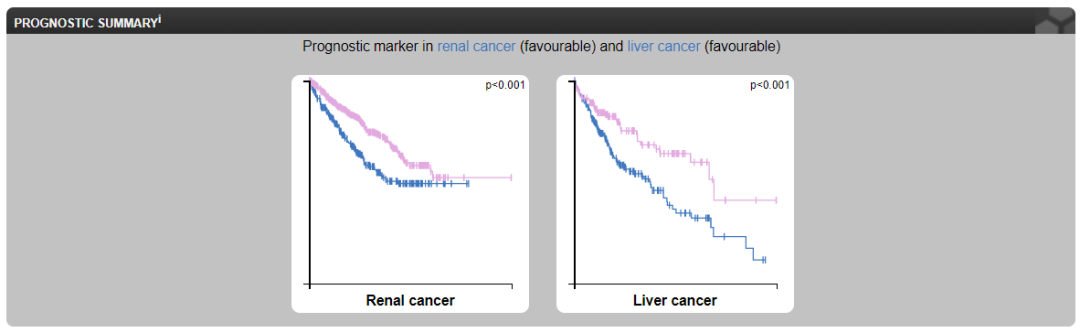
在肿瘤中的表达,没有展示对照组不利于结果的呈现。所以需要借助ONCOMINE数据库的数据。
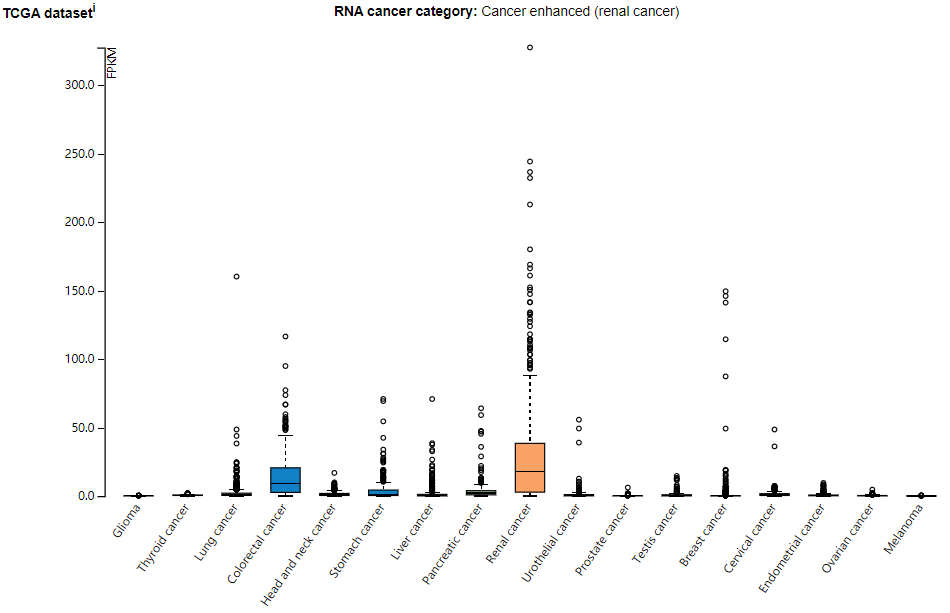
在肿瘤中组织中表达的情况总结。
在脑中的表达。
在各种血细胞和血浆中的表达。

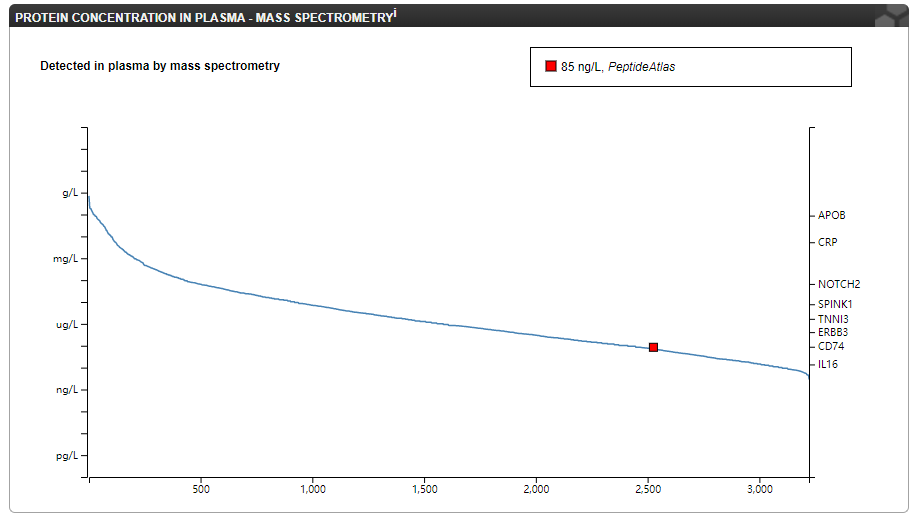
质谱技术检测
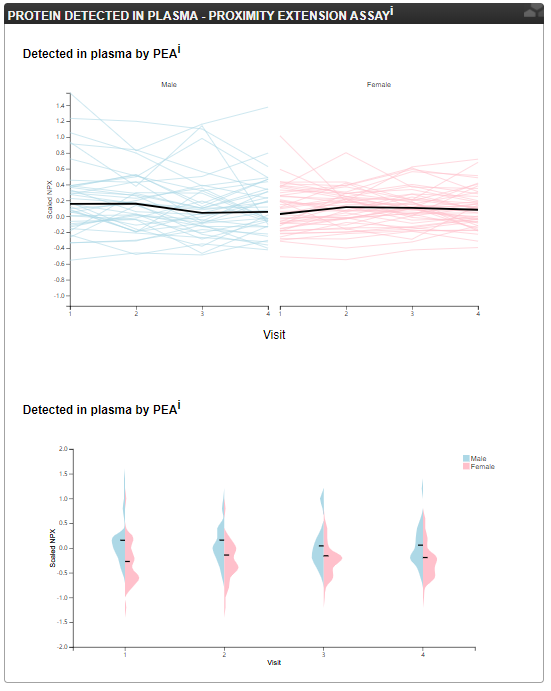
PEA分析
那么,关键问题来了,如何用HPA数据库的数据,发表论文呢?
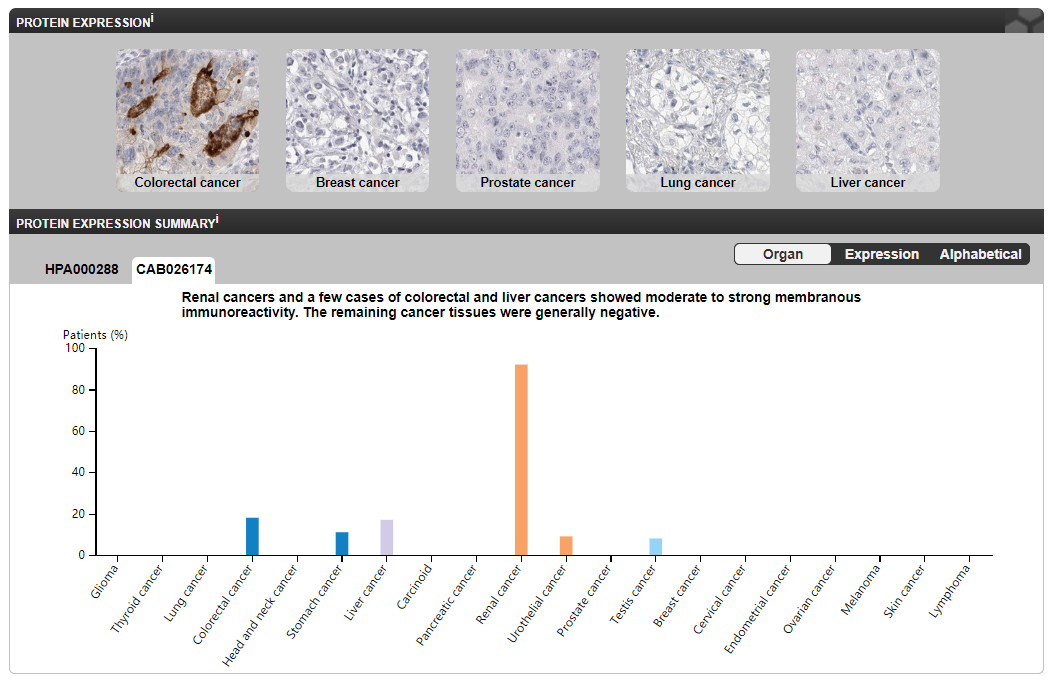
一般在论文中出现的是病理检测结果(IHC),特别是结合ONCOMINE数据库的结果,对基因在肿瘤和临近组织的表达进行对比展示。在前面的文章中,我们介绍过。结果展示如下。

具体操作如下。
打开病理那个界面。
然后根据病人信息,选取具代表性的样本,放至PPT中,进行展示。

同时,HPA提供患者的信息,根据患者信息,还可以做出回归曲线。
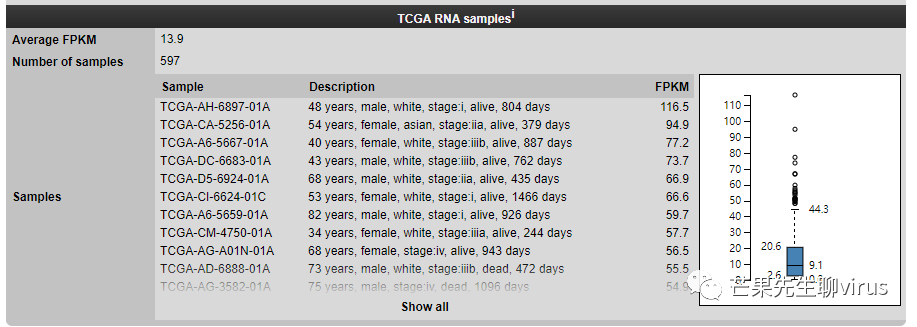
3. 对照病理组织的结果在Tissue里面,放至PPT中,与肿瘤的病理报告放在一起,对比展示。这样就大功告成了。
本文分享自微信公众号 - 芒果先生聊生信(from2019to2020)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。