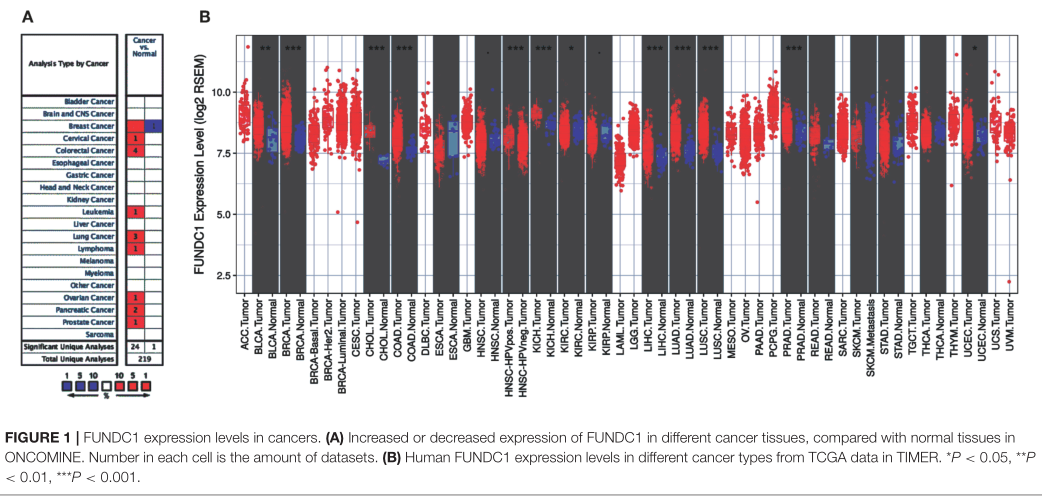
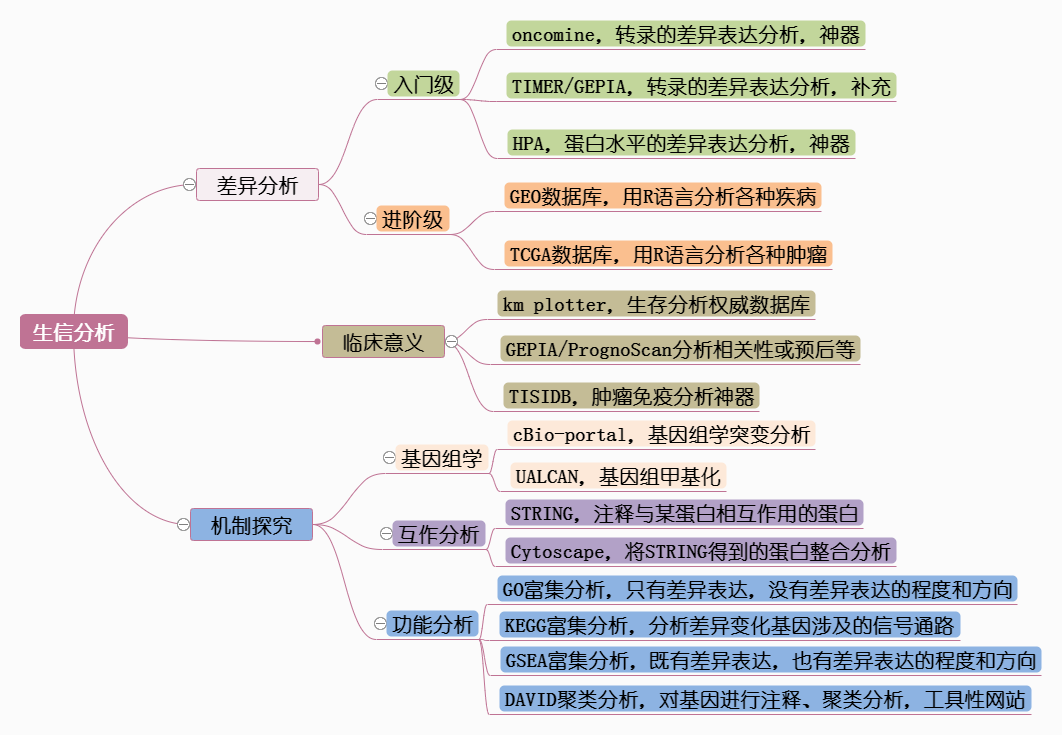
生信论文的套路
ONCOMINE从全景、亚型两个维度做表达差异分析;
临床标本从蛋白水平确认(或HPA数据库),很重要;
Kaplan-Meier Plotter从临床意义的角度阐明其重要性;
cBio-portal数据库做基因组学的分析(机制一);
STRING互作和GO/KEGG分析探讨可能的信号通路(机制二);
TISIDB/TIMER分析肿瘤免疫特征(机制三)。
前面,我们已经分享oncomine数据库做转录水平的差异分析,并推荐用oncomine+GEPIA双验证模式做差异分析。鉴于最新发表的论文和肿瘤浸润免疫细胞表型的分析,个人认为oncomine+TIMER双验证也是不错的组合。
对于单基因的差异分析,尤其是与肿瘤浸润免疫细胞表型相关的分析,芒果建议采用这种方法,确实做到统筹兼顾,有局部聚焦(oncomine)和全局通览(TIMER)的神奇效果。不过,在做多基因或者家族基因分析的时候,是选择oncomine+GEPIA双验证模式,而是选择oncomine+TIMER双验证,可以具体问题具体分析。
生信分析36
生信分析37
生信论文38
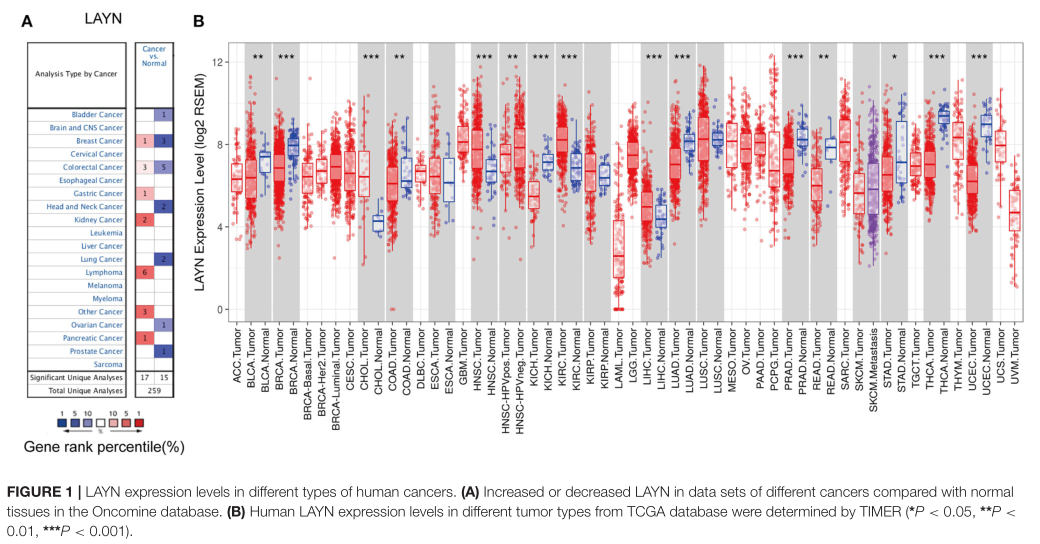
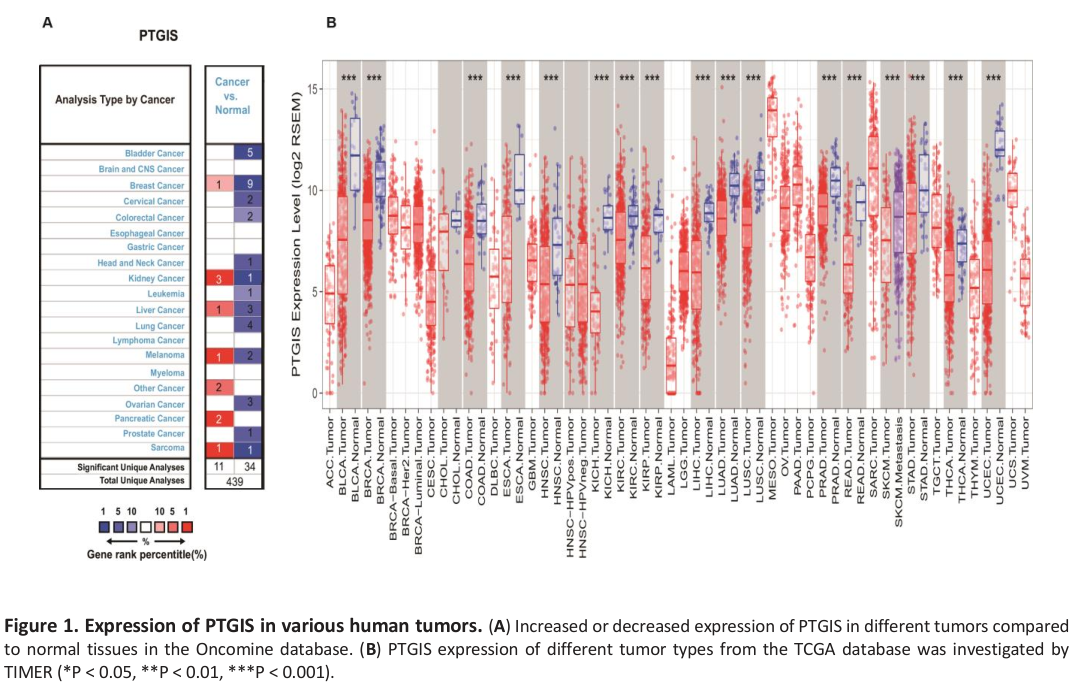
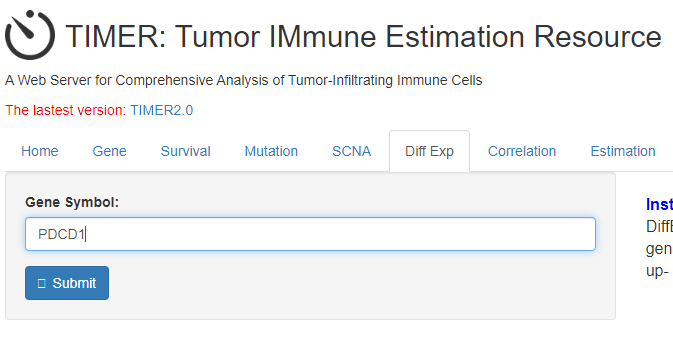
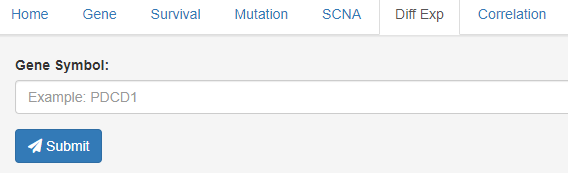
TIMER数据库也是可以做差异表达分析的,而且还不错呢。在Diff Exp选项输入基因名称,点击submit即可生成。
TIMER (Tumor Immune Estimation Resource)数据库也是用高通量测序(RNA-Seq表达谱)数据分析肿瘤组织中免疫细胞的浸润情况,主要提供B cells, CD4+ T cells, CD8+ T cells, Neutrphils, Macrophages and Dendritic cells等六种免疫细胞的浸润情况。界面友好,简单易学又方便。网址:https://cistrome.shinyapps.io/timer/。
TIMER网站分7个模块,其中前6个模块是对TCGA数据库的分析展示,第7个模块是对免疫细胞浸润比例进行评估。

基因模块,输入基因、肿瘤类型后,submit即可。

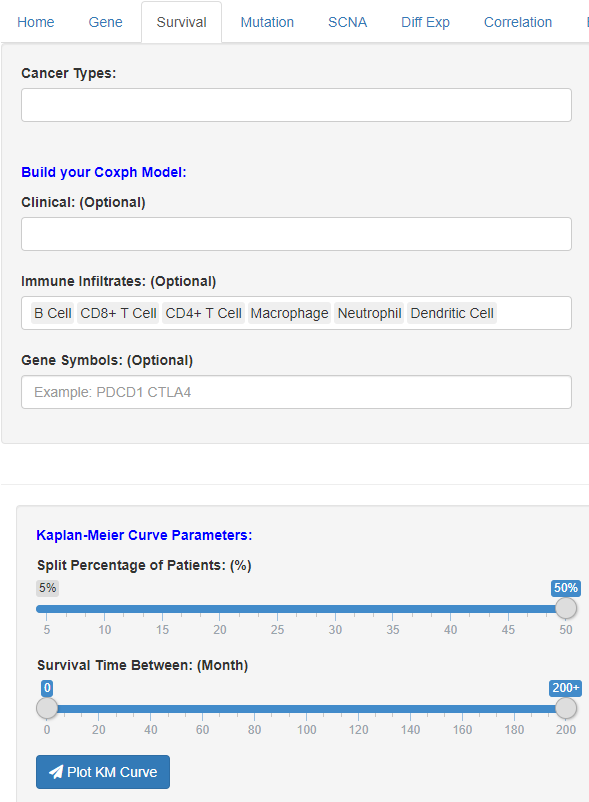
存活率模块。设定肿瘤类型,临床参数,免疫细胞类型和基因,还可以设定患者比例,存活时间,各种组合在一起,可以出很多图和表。功能强大。
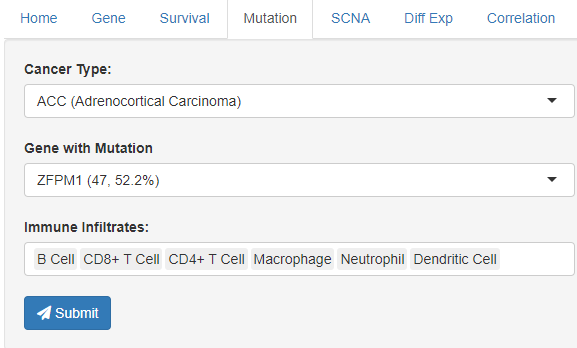
突变模块,此部分内容相对局限,只有部分常见的突变基因列出。
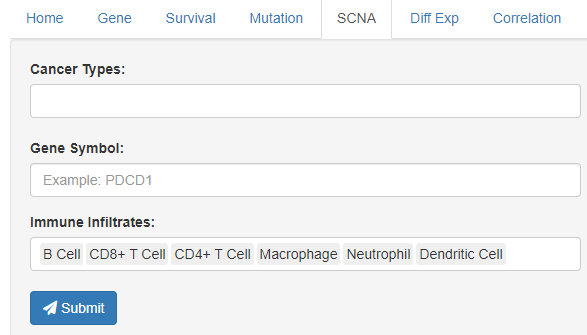
SCNA模块,肿瘤和基因输入,submit即可。
差异表达分析。
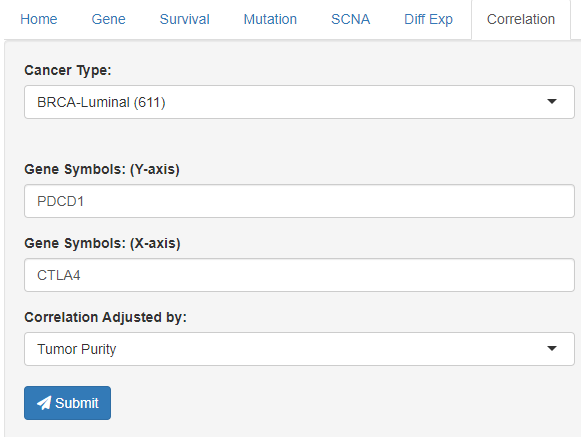
相关性分析。
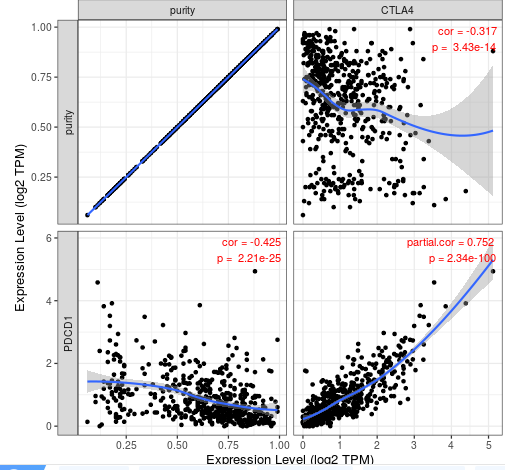
最后的评估模块其实是该数据库的特色。在掌握下载TCGA数据路数据的条件下,结合该数据库的这种功能,接近更高层次的论文。
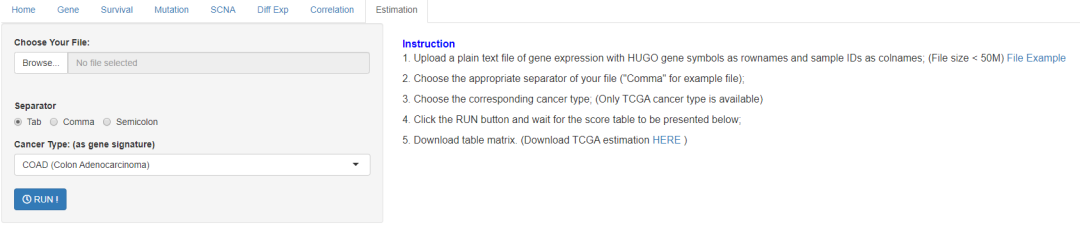
比较起来,其实TIMER比TISIDB更简单,因为TISIDB的数据需要判断后选择可用的数据。在进行肿瘤免疫浸润分析时,最好能多角度分析。
本文分享自微信公众号 - 芒果先生聊生信(from2019to2020)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。