最近有朋友和我交流纳米孔16S测序数据的分析,发现真的没有从头完成过一次这方面的数据分析,然后发现这方面的资料也比较少,于是学习一下,和大家分享。坦白说,牛津纳米孔测序技术在16S多样性研究方面还是有些不足的,只能说勉强够用(准确性大概也不能完全保证),主要应用场景是在一些现场快速检测方面,主要是病原菌这种。但是,相信随着测序准确度的提高和分析软件的改进,相信它的应用会越来越多。感谢互联网的便利和分享精神,今天的我们可以方便地获得测序的原始数据,并可以自由进行分析。
last方面的内容主要参考自IT帮一个兄弟的博客,之前提到过[1]。
1.软件和数据准备
处理牛津纳米孔的测序数据,首先当然要安装相关的专用软件了,基本上原厂出的,原厂品质,放心!还有就是minimap2和yacrd,用于去嵌合。数据来自一篇文章,文件不大,直接下载或者ascp下载均可。
#这个流程可参考https://github.com/tetedange13/stage_M2BI_scripts#软件准备#首先是NanoPlot,用于检测测序质量,直接pip安装了,加速的话可使用清华源pip install NanoPlot #去接头的Porechopgit clone https://github.com/rrwick/Porechop.gitcd Porechoppython3 setup.py installporechop -h #质控的NanoFilt,pip或bioconda安装pip install nanofilt#minimap2,编译或者下载预编译的二进制文件均可,这里编译git clone https://github.com/lh3/minimap2cd minimap2 && make#yacrd,conda安装conda install yacrd#获取数据,我是ascp下载/Volumes/10.11/Users/zd200572/Applications/Aspera\ Connect.app/Contents/Resources/ascp -i ~/asperaweb_id_dsa.openssh -Tr -Q -l 100M -P33001 -L- fasp-g1k@fasp.1000genomes.ebi.ac.uk:vol1/fastq/ERR277/007/ERR2778177/ERR2778177.fastq.gz .#比对工具last,一款来自日本的比对软件,需要编译wget http://last.cbrc.jp/last-1060.zipunzip last-1060.zipcd last-1060make#下载相应版本centrifuge,解压wget ftp://ftp.ccb.jhu.edu/pub/infphilo/centrifuge/downloads/centrifuge-1.0.3-beta-Linux_x86_64.zipunzip centrifuge-1.0.3-beta-Linux_x86_64.zip#如果wget或者浏览器下载地址是wget http://ftp.ebi.ac.uk/vol1/fastq/ERR277/007/ERR2778177/ERR2778177.fastq.gz#ncbi的16S数据库,由于出国网络差,下了好久,一定要确保数据下载完整,注意下载时勾选gi号,以备后面有用#https://www.ncbi.nlm.nih.gov/nuccore?term=33175%5BBioProject%5D#这里我把这个文件存在了网盘,公众号内回复“16S”即可获得下载地址
2.数据预处理
基本上是质控的过程,先看下测序质量,当然纳米孔的质量还是有点低的,特别是手上下载的数据是低版本的测序芯片R9.4,未来的R10可以通过两个纳米孔串联提高到95%。接着就是去除测序的接头,获得真正的测序序列,是不是引物也应该切除?但是估计数据库里的序列也应该不包含引物,所以估计引物影响不大。然后,进行过滤,除去明显不符合要求的序列。
#查看质量,fastq最常用,这里用官方的试试NanoPlot -t 4 --fastq ERR2778177.fastq.gz --plots hex dot
结果依然是类似于fastq质控报告的一个函数,不过统计指标少了几个,有两个把reads长度和质量分布放在一起的图不错。
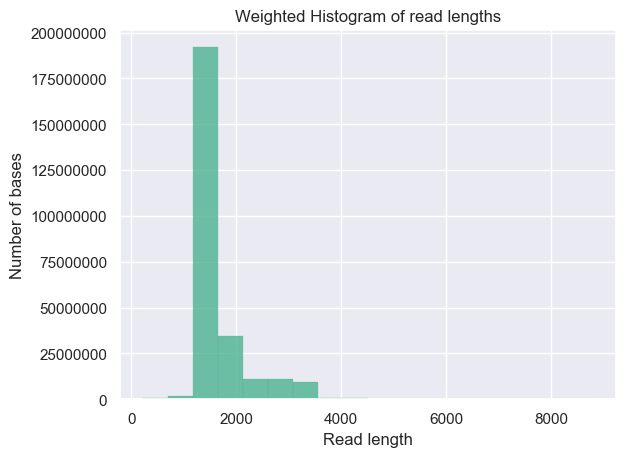
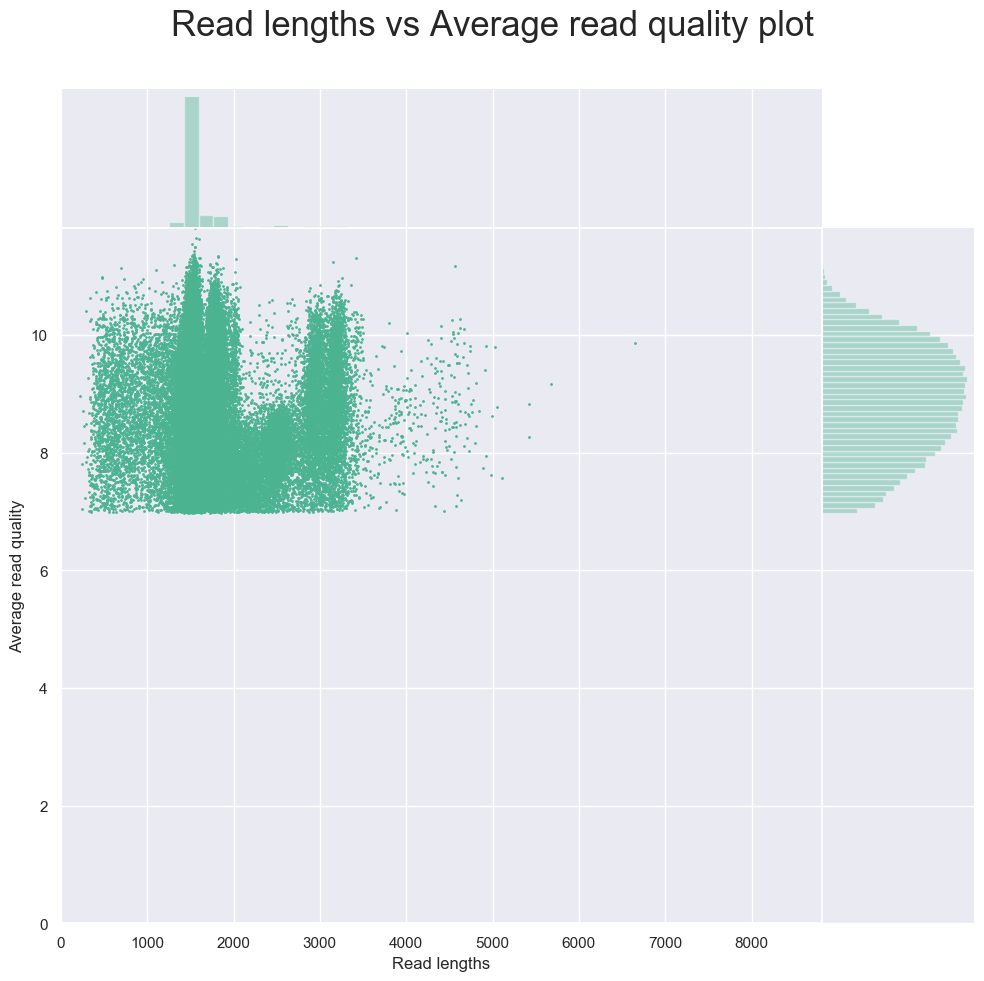
img
#去接头,4线程,这一步耗时以小时计,对于我的五年前的双核心四线程笔记本 porechop -t 4 -i input.fastq -o trimmed.fastq# 质控,质量值为9,长度200过滤,虽然有点低,这有两个重定向NanoFilt -q 9 -l 200 < trimmed.fastq > cleaned.fastq#去嵌合,先minimap2自全局比对,发现嵌合,然后yacrd去除minimap2/minimap2 -x ava-ont -g 500 cleaned.fastq cleaned.fastq > overlap.pafyacrd -i overlap.paf -o report.yacrd -c 4 -n 0.4 scrubb -i cleaned.fastq -o reads.scrubb.fq#卡在这时间最长,主要几个文件要准备,需要整理的已经放在文件夹,其余的可以ncbi下载,较大,没放,或者直接用我建好的。#看下文件的保留情况,大概去除了一半以上的数据tree -h | grep q├── [237M] ERR2778177.fastq.gz├── [224M] cleaned.fastq├── [200M] reads.scrubb.fq└── [476M] trimmed.fastq
3. last比对
关于纳米孔16S的数据分析,之前翻译的那篇综述总结了大概有两种,一种是和之前的16S数据分析一样聚类ASV/OTU,但是由于90%左右的准确度,看有用85%的准确度聚类的。。。另一种就是,不聚类,直接进行比对,也就是我们这次学习的blast等比对工具的方法,根据综述作者的观点,这几种工具由于不是专门为纳米孔测序数据设计,不能比较好的完成物种注释的任务(不够准确),作者推荐的是minimap2和centrifuge这两个软件。但是,有很大一部分文章是以这几个工具进行数据分析的,我们也做一下,最后进行一个比较吧!
# 构建参考数据库last-1060/src/lastdb -uYASS -R01 microbialdb bacteria.16SrRNA.fna# 比对https://gigascience.biomedcentral.com/articles/10.1186/s13742-016-0111-z#Sec12#取前10000序列测试,减少运行时间
4.结果处理,获得物种丰度表
由于自己水平还不够写脚本从比对结果中获得物种注释,脚本基本上来自台湾的那个同仁的。确切的说应该还是对比对结果的数据结构不够熟悉,争取后面多熟悉些。为了实现使用他的脚本,对数据库的信息进行了一些小的提取操作,用我暂时用得比较顺手的python完成的。我已经把处理好的数据上传了百度云,直接使用的话可以略过。
--------------我是分界线的开始-----------
cat ncbi-16S.fasta | grep ">" > conv.txt #先获得序列名信息备用
dic_fa = {}i = 0with open('conv.txt') as f: for line in f: i += 1 if line.strip() != '': ac_n = line.strip().split('|')[3] gi_n = line.strip().split('|')[1] dic_fa[ac_n] = '' dic_fa[ac_n] = [gi_n, line.strip()]print(i)j = 0fout = open('info.txt', 'w')with open('sequence.gb') as f1: for line in f1: if line[:7] == "VERSION": seq_name = line.strip().split(' ')[1].split(' ')[0] if '/db_xref="taxon:' in line: tax_id = line.strip().split('/db_xref="taxon:')[1].split('"')[0] if seq_name in dic_fa.keys(): fout.write(dic_fa[seq_name][1] + '\t' + tax_id + '\n') #+ seq_name + '\n') seq_name = '' tax_id = '' j += 1print(j)fout.close()
--------------我是分界线的结束-----------
# 加载包,第一次看到这样的方式suppressMessages(library(data.table))suppressMessages(library(tidyverse))# 设置文件位置id_mapping_file <- "info.txt"blastOutputs <- "aligned.txt"#导入输入文件id_mapping <- fread(id_mapping_file, header = FALSE, sep = "\t", stringsAsFactors = FALSE)setkey(id_mapping, V1)blastoutput <- fread(blastOutputs, header = FALSE, sep = "\t", stringsAsFactors = FALSE)blastoutput_filtered <- blastoutput %>% dplyr::filter(V3>=90 & V4>=700) %>% group_by(V1) %>% dplyr::filter(V3 == max(V3)) %>% dplyr::filter(V12 == max(V12))allreadsnum <- length(unique(blastoutput$V1))readStats <- table(blastoutput_filtered$V2)refseqIds <- names(readStats)df <- data.frame(RefseqID = refseqIds, Lineage = NA, ReadsNum = NA, OrgName = NA, ReadsPerc = NA)#循环获得信息for (i in 1:length(refseqIds)) { refseqid <- refseqIds[i] tmp <- id_mapping[.(refseqid)] lineage <- tmp$V3 orgname <- tmp$V4 readnum <- readStats[refseqid] perc <- readnum*100/allreadsnum df$Lineage[i] <- lineage df$ReadsNum[i] <- readnum df$ReadsPerc[i] <- perc df$OrgName[i] <- orgname}taxonomy <- paste(df$Lineage, df$OrgName, sep="; ")output_df <- data.frame(Taxonomy = taxonomy, ReadsNumber = df$ReadsNum)output_df <- aggregate(output_df$ReadsNumber, by = list(Taxonomy=output_df$Taxonomy), FUN = sum)colnames(output_df)[2] <- "ReadsNumber"ReadPercentage <- output_df$ReadsNumber*100/allreadsnumoutput_df <- cbind(output_df, ReadPercentage)output_df <- output_df[order(output_df$ReadPercentage, decreasing = TRUE),]#output <- paste0("tax_", sub("\\.\\/Alignment\\/", "", file))write.table(output_df, 'out.txt', sep = "\t", row.names = FALSE, quote = FALSE)
最后,如果顺利,就是结果了:
Taxonomy
ReadsNumber
ReadPercentage
Trichocoleus desertorum strain ATA4-8-CV2
1046
41.85674270
Neosynechococcus sphagnicola strain sy1
248
9.92396959
Tychonema bourrellyi strain CCAP 1459/11B
110
4.40176070
Kastovskya adunca strain ATA6-11-RM4
104
4.16166467
Okeania plumata strain FK12-27
74
2.96118447
Loriellopsis cavernicola strain LF-B5
53
2.12084834
Cephalothrix komarekiana CCIBt 3277
42
1.68067227
Caedimonas varicaedens strain 221
41
1.64065626
Aliterella antarctica strain CENA408
33
1.32052821
Limnoraphis robusta strain CCALA 966
31
1.24049620
5. centrifuge物种注释
卡在这时间最长,主要几个文件要准备,需要整理的已经放在文件夹,其余的可以ncbi下载,较大,没放,或者直接用我建好的,公众回复16S 即可获得下载地址。
#build 10min,这步可以不用进行,已经有构建好的文件 ncbi_16s#./centrifuge-1.0.3-beta/centrifuge-build -p 4 --bmax 1342177280 --conversion-table info.txt \# --taxonomy-tree /Volumes/LENOVO_imcdul/db/NCBI_tax/nodes.dmp --name-table /Volumes/LENOVO_imcdul/db/NCBI_tax/names.dmp \# reference.fasta ncbi_16s#注释../Help/centrifuge/centrifuge test.fq -x ../Help/ncbi_16s -p 4 -t -S log.txt../Help/centrifuge/centrifuge-kreport -x ../Help/ncbi_16s centrifuge_report.tsv > test.txt
试了下发现两者的差别还是比较大的,这意味着很大的不可靠性,如果有新的方法和思路,欢迎交流,共同进步。之前nano-ampliseq[2]的流程就是因为没仔细阅读脚本说明,没有充分准备好依赖条件和软件,导致没有结果,感谢有同仁指出!阅读原文是我修正了部分内容的那个流程,时间关系,没有跑一遍。
其他可用的流程还包括同仁提到的q2-ONT流程(我试用下来物种注释结果相当不理想,难道是85%聚类的原因),以及那篇综述提到的聚类方法,总的来说,分析方法还是不够成熟,还需要继续改进下去!
前面提到的几个文件的分享链接: https://pan.baidu.com/s/1FOp6kU2dB\_37\_Wi\_mH2F0Q 提取码: 8gce
[1]https://ithelp.ithome.com.tw/articles/10200625
[2] https://blog.csdn.net/zd200572/article/details/94575020
本文分享自微信公众号 - 科技记者(kejijizhe)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。