版权声明:本文为博主原创文章,遵循 CC 4.0 BY-SA 版权协议,转载请附上原文出处链接和本声明。
本文链接: https://blog.csdn.net/devcloud/article/details/94549627
GO是Gene Ontology的简称,是基因功能国际标准分类体系。它旨在建立一个适用于各种物种的,对基因和蛋白质功能进行限定和描述的,并能随着研究不断深入而更新的语言词汇标准。GO分为分子功能(Molecular Function)、生物过程(Biological Process)、和细胞组成(Cellular Component)三个部分。
富集分析主要用于差异基因在GO term的富集程度,颜色越深富集越显著,红色最显著,黄色次之,无色代表富集不显著。
- GO term分为三大类,每一类从不同的层面解释基因的生物学功能,我们可以结合生物学问题的特殊性,有针对性的关注GO term:例如我们期望从离子通道这一层面解释植物耐旱,耐盐的的机理,我们可以优先关注细胞组成里面膜蛋白。
- GO term间具有包含关系,GO term之间可以构建复杂的结构网络。GO term 层级越低,功能描述越具体,越是低层级,越能解释生物学的问题,所以我们要关注显著富集的低层级GO term,以便具体而详尽的解释生物学问题。
- GO富集分析的统计假设,并不能完全代基因功能的重要程度。要结合生物学问题、结合基因的功能注释,才能判断其中的基因变化是否有重要的生物学意义。
这里可以使用clusterProfiler找到富集的GO
安装所需的R包
1 source("https://bioconductor.org/biocLite.R")
2 options(BioC_mirror="http://mirrors.ustc.edu.cn/bioc/")
3 biocLite("org.Hs.eg.db")
4 biocLite("clusterProfiler")
5 install.packages("ggplot2")
进行富集分析
1 library(org.Hs.eg.db)
2 library(ggplot2)
3 setwd("D:/medical_service/go_enrich")
4 # geneNames <- c("AHNAK2", "AQP7", "DNAH11" , "FLG", "HNRNPCL2", "HRNR" , "KMT2C",
5 # "KMT2D", "MST1L", "MUC12", "MUC16", "MUC17", "MUC19", "MUC3A",
6 # "MUC4", "MUC5B", "MUC6", "PABPC3", "PDE4DIP", "PLEC" , "TTN",
7 # "ANKRD36", "FCGBP", "HERC2", "IGFN1", "KRT18", "SLC25A5", "SYNE2",
8 # "RYR1", "TNS1", "DST", "SYNE1", "TSNARE1", "NBPF19", "NBPF26",
9 # "PRKCB", "ADGRG1", "OPCML")
10 d1 <- read.table("genenames.txt", header=T, stringsAsFactor =F)
11 geneNames <- d1$GeneName
12 gene <- mapIds(org.Hs.eg.db, geneNames, 'ENTREZID', 'SYMBOL')
13 BP.params <- enrichGO( gene = gene,
14 OrgDb = org.Hs.eg.db,
15 ont = "BP" ,
16 pAdjustMethod = "BH",
17 pvalueCutoff = 0.01,
18 qvalueCutoff = 0.05)
19
20 BP.list <- setReadable(BP.params, org.Hs.eg.db, keyType = "ENTREZID")
21
22 dotplot(BP.list, showCategory=30)library(clusterProfiler
如果要做BP, CC, MF的综合柱状图,采用ggplot2
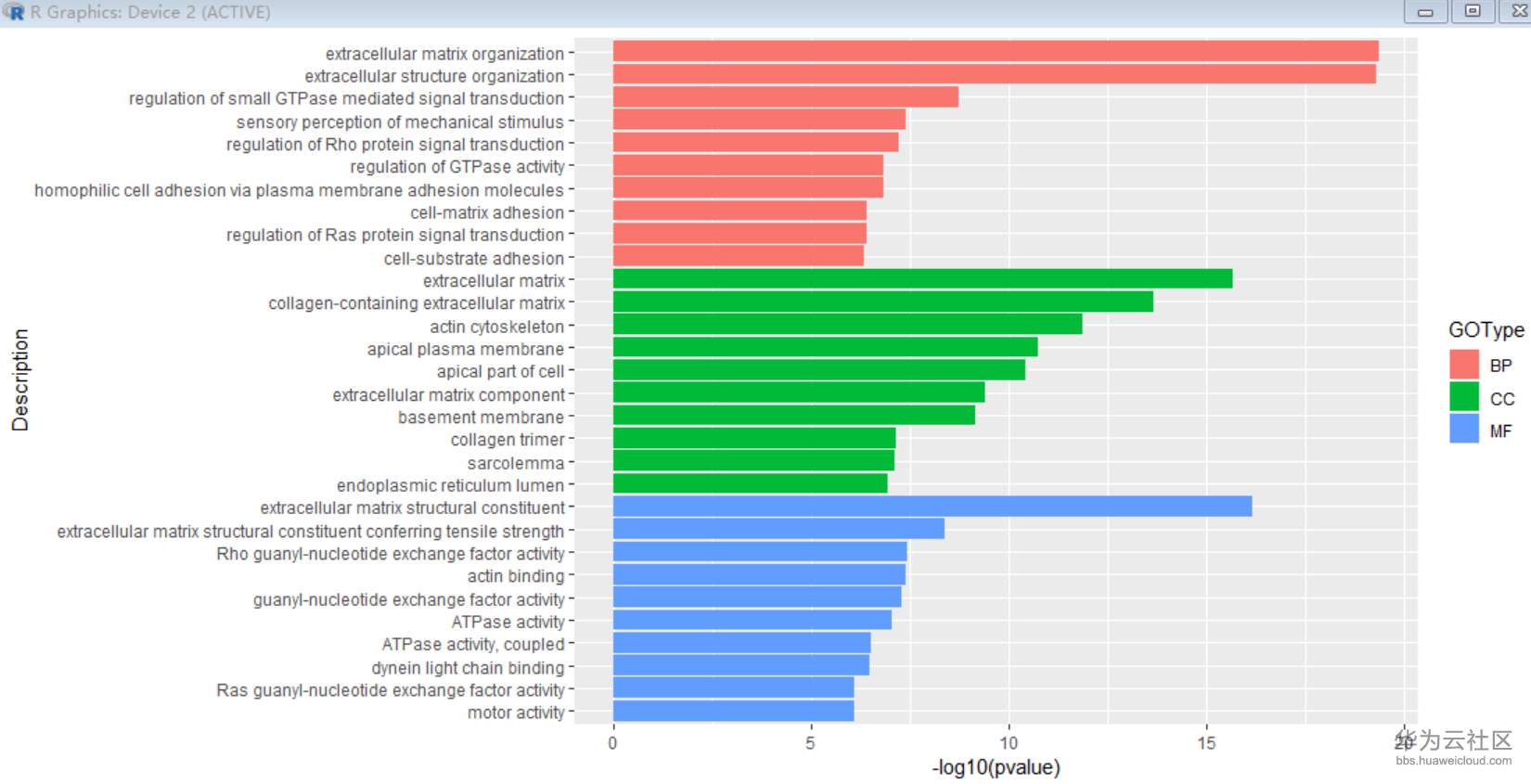
1 p1 <- ggplot(data=goAll)+ geom_bar(aes(x=Description,y=-log10(pvalue), fill=GOType), stat='identity') + coord_flip() + scale_x_discrete(limits=goAll$Description)
2
3 ggsave("out_bar.pdf", p1, width = 10, height=6)
4
5
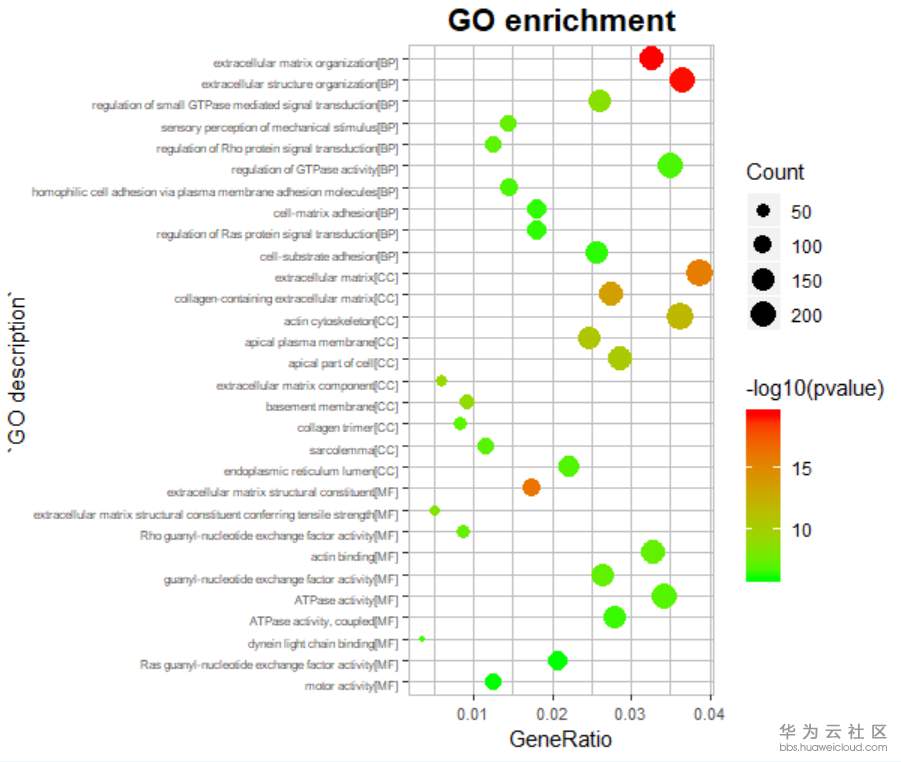
6 p2 <- ggplot(Edata, aes(x=GeneRatio, y=`GO description`)) +
7 geom_point(aes( size= Count , colour = -log10( pvalue )) ) + scale_y_discrete(limits=Edata$`GO description`)+
8 ggtitle("GO enrichment") + scale_color_gradient(low = 'green', high = 'red') + xlim(range(Edata$GeneRatio)) +
9 theme(axis.text.x=element_text(angle=0,size=8, vjust=0.7), axis.text.y=element_text(angle=0,size=6, vjust=0.7),plot.title = element_text(lineheight=.8, face="bold", hjust=0.5, size =16), panel.background = element_rect(fill="white", colour='gray'), panel.grid.major = element_line(size = 0.05, colour = "gray"), panel.grid.minor.y = element_line(size=0.05, colour="gray"), panel.grid.minor.x = element_line(size=0.05, colour="gray")
10 )
11
12 ggsave("out_GO.pdf", p2, width = 8, height=7)
效果如图
来源:华为云社区 作者:benymorre
HDC.Cloud 华为开发者大会2020 即将于2020年2月11日-12日在深圳举办,是一线开发者学习实践鲲鹏通用计算、昇腾AI计算、数据库、区块链、云原生、5G等ICT开放能力的最佳舞台。







