注意:本文的内容主要来自于GATK官网的讲解,所以vcf也是GATK产生的,用其他caller,比如varscan2产生的vcf文件的内容注释可能不一致。
VCF:由HEADER和RECORDS组成。
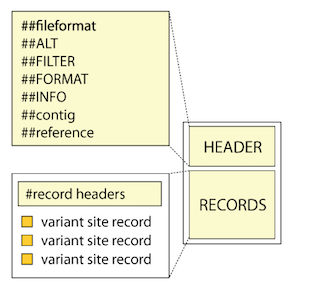
RECORDS的FORMAT内容详解:
QUAL:指的是caller正确的识别该变异位点的可能性,属于phred-scale quality score的一个应用。
GT,GQ,PL三者的关系:
GT是指该位点最有可能的基因型。
GQ是该位点第二有可能的基因型的PL值。
PL是不同基因型对应的标准化的可能性。
对于二倍体生物来说,PL有三个值,分别对应0/0,0/1,1/1。最有可能的基因型的PL值为0,第二小的是第二个可能。GQ反映的是第二个小的基因型的PL值,如果该值超过99,则定位99,因为超过了99,其几乎不能威胁第一个可能的地位。
计算方法:
PL(0/1) = 0 (the normalized value that corresponds to a likelihood of 1.0) as is always the case for the assigned allele,
but the next PL is PL(1/1) = 26 (which corresponds to 10^(-2.6), or 0.0025).
QUAL和GQ的区别:

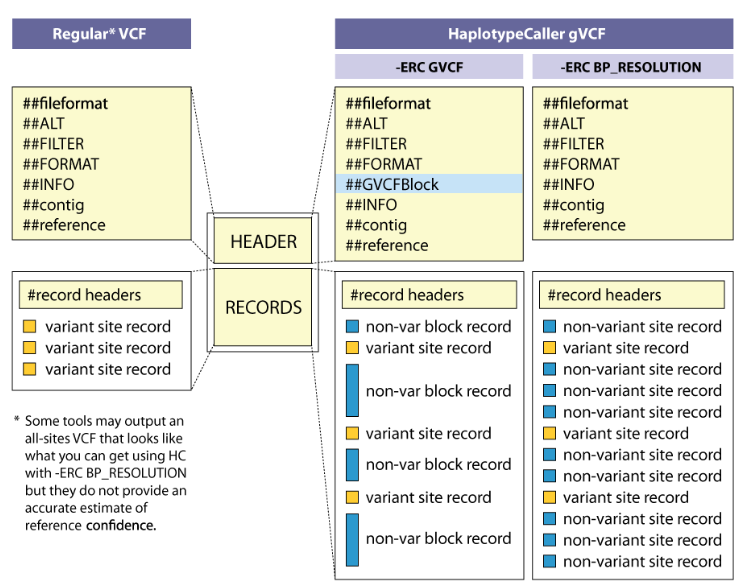
GVCF和VCF的最大区别是在于GVCF文件会记录所有的点,包括哪些没有突变的点。
在GVCF模式下,那些没有变异的点会形成一个未变异块,non-var block record。
GVCF的好处:能更方便把一群样本的GVCF联合起来,以便进行下一步分析,提高分析效率。而且GVCF的records还会提供一个
The records in a gVCF include an accurate estimation of how confident we are in the determination that the sites are homozygous-reference or not. This estimation is generated by the HaplotypeCaller's built-in reference model