1.supplementary alignment
supplementary alignment是指一条read的一部分和参考区域1比对成功,另一部分和参考区域2比对成功,参考区域1和参考区域2没有交集(或很少),那么一条read就会产生两条sam文件,
将其中的一条sam文件作为represent alignment,而另一条作为supplementary alignment (flag为2048)。
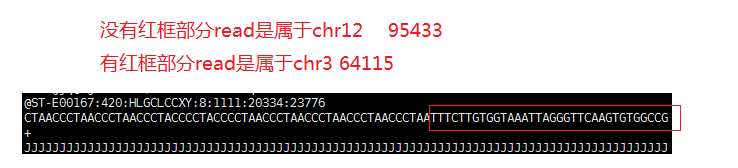
将上面的fastq文件去跑bwa,read有两条sam文件,第二条的flag值为2048:

2.bwa mem的-M -Y参数:
-M:mark shorter split hits as secondary。就是把supplemenary alignment 变为no primary(flag值256) 。下面是bwa mem -M的结果

-Y:use soft clipping for supplementary alignments。把默认的hard clip变为soft clip。hard clip 不会显示不匹配的碱基串,soft clip会显示不匹配的碱基串。下面是bwa mem -Y的结果(58H34M变为58S34M)

3.secondary(no primary)是指这条read在基因组上有多个匹配区域(>=2),可以是read是的同一部分有不同匹配区域,也可以是一条read上的不同区域。所以supplemenary aligment应该算是secondary的子集。
许多处理bam的软件不会去处理supplemenary(split alignments),比如Picard’s markDuplicates,所以可能需要用-M把supplemenary 转换为secondary。