点击上方“蓝字”带你去看小星星
Y叔开发的 ChIPseeker包,主要是为了能 对ChIP-seq数据进行注释与可视化,主要对peak位置及peak邻近基因的注释。然而,在之后对ChIPseeker的应用中,发现它不局限于ChIP-seq,可用于其他的peak(如ATAC-seq,DNase-seq等富集得到的)注释,甚至还可用于long intergenic non-coding RNAs (lincRNAs)的注释。该包功能强大之处还是在于可视化上。
这里我们主要介绍ChIPseeker包用于ChIP-seq数据的注释与可视化,主要分为以下几个部分。

0 1
数据准备
在用ChIPseeker包进行注释前,需要准备两个文件:
1
注释peaks的文件
该文件需要满足BED格式。BED格式文件至少得有chrom(染色体名字),chromStart(染色体起始位点)和chromEnd(染色体终止位点),其它信息如name,score,strand等可有可无。一般情况而言,可直接用做peak calling的MACS输出文件(以_peaks.bed结尾文件)。
2
有注释信息的TxDb对象
Bioconductor包提供了30个TxDb包,包含了很多物种,如人,老鼠等。当所研究的物种没有已有的TxDb时,可利用GenomicFeatures包进行制作:
makeTxDbFromUCSC:通过UCSC在线制作TxDb
如有对应物种的gff文件,用makeTxDbFromGFF来制作TxDb:
require(GenomicFeatures)

0 2
数据可视化
查看peak在全基因组的分布情况,用covplot可视化BED格式文件:
##安装并加载ChIPseeker包
> files
files包括5个BED格式文件,对应不同样本数据。
##可视化第4个BED格式文件

同时可视化多个BED格式文件:
require(GenomicFeatures)
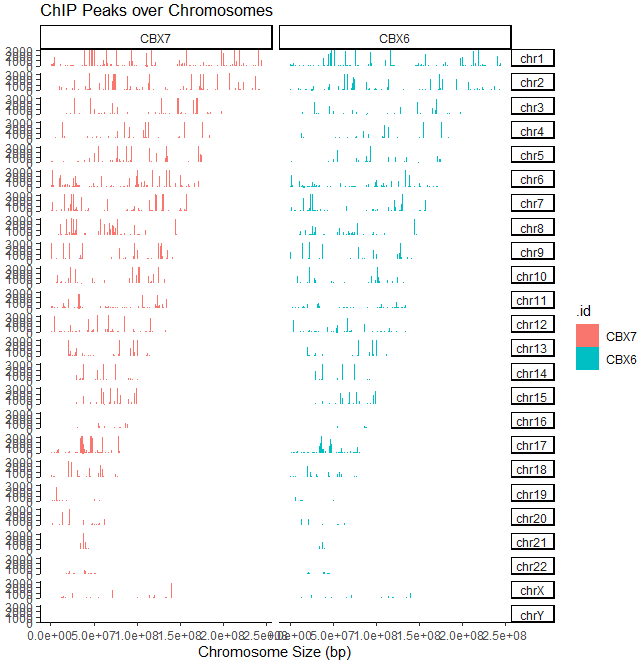
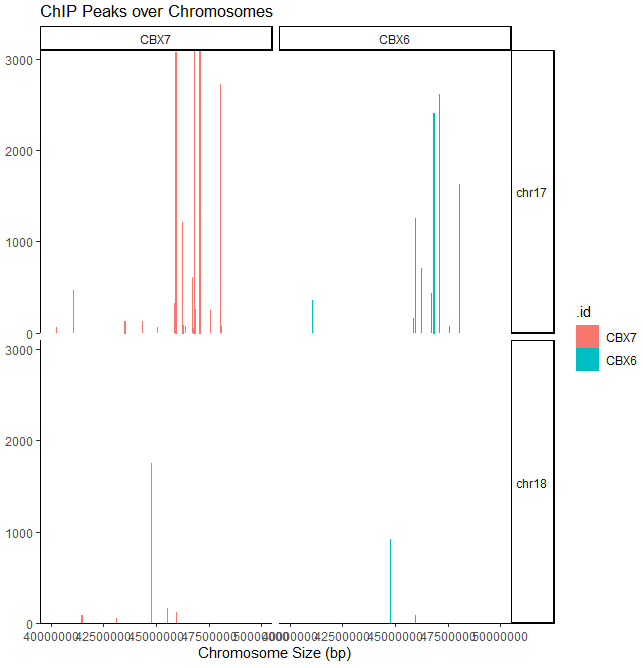
只关注其中的几条染色体:
covplot(peak, weightCol="V5", chrs=c("chr17", "chr18"), xlim=c(4e7, 5e7)) + facet_grid(chr ~ .id)

0 3
Peak可视化
tagHeatmap函数查看TSS(转录起始位点)上下游3kb区域peak的分布情况:
##加载对应的TxDb包
##getPromoters函数准备启动子窗口
##getTagMatrix函数把peaks比对到启动子窗口上
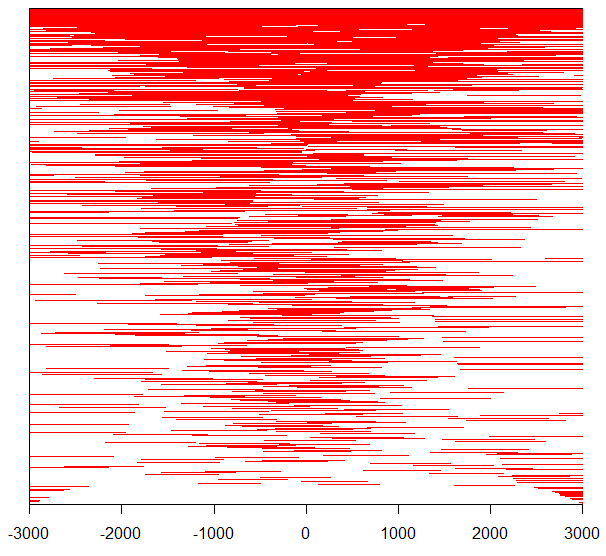
用peakheatmap函数查看多个样本TSS上下游3kb区域内peak的分布情况:
peakHeatmap(files, TxDb=txdb, upstream=3000, downstream=3000, color=rainbow(length(files)))
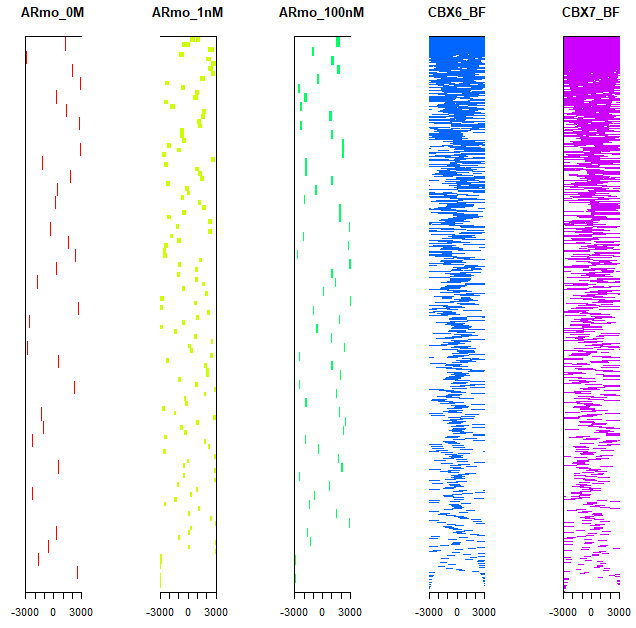
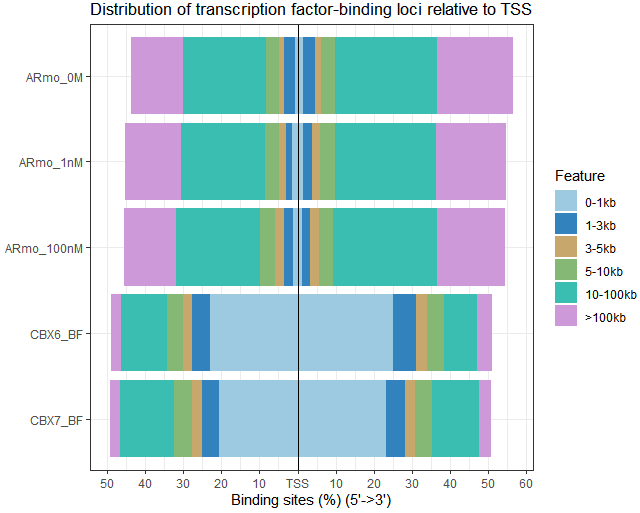
这里我们可以看出ARmo_0M,ARmo_1nM和ARmo_100nM三个样本的结合位点不在TSS附近,而CBX6_BF和CBX7_BF两个样本结合位点在TSS附近。
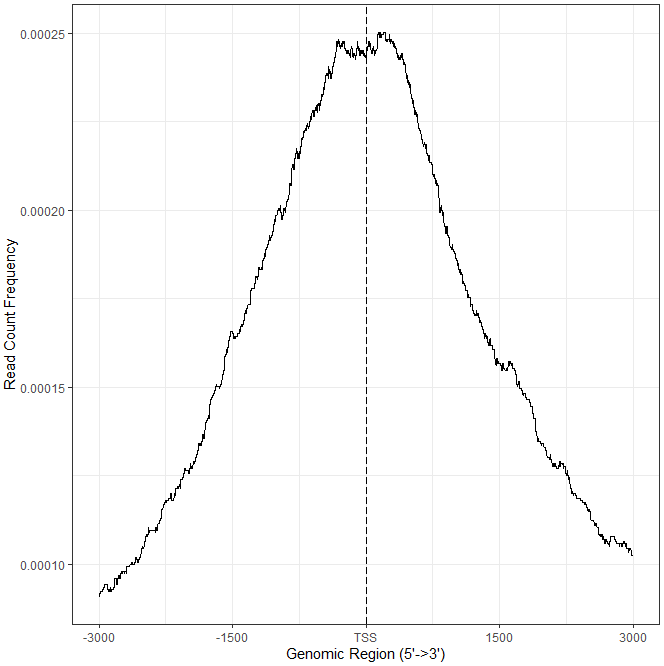
用plotAvgProf函数查看结合的强度:
plotAvgProf(tagMatrix, xlim=c(-3000, 3000),xlab="Genomic Region (5'->3')", ylab = "Read Count Frequency")
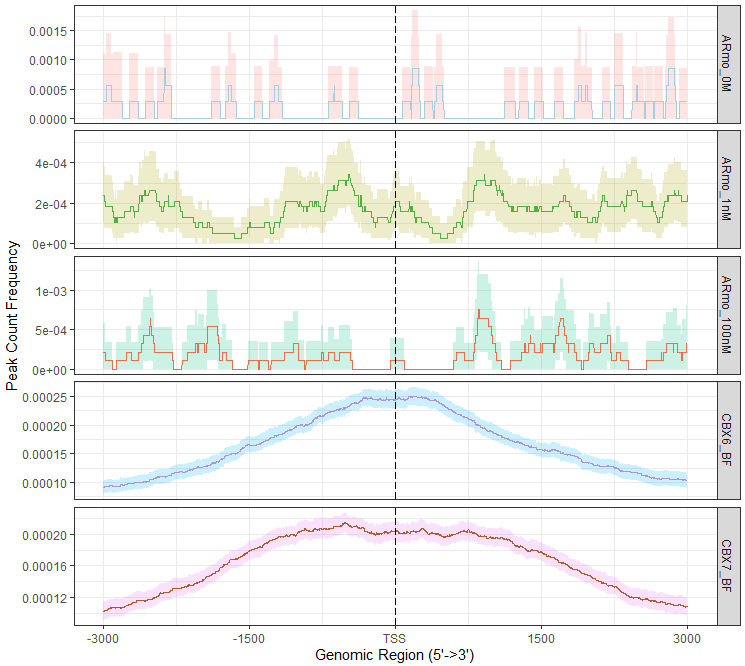
plotAvgProf 函数同时查看多个样本结合的强度:
##lapply函数进行批量处理

0 4
peak注释
这里用annotatePeak函数来对peak进行注释(准备好前面那两个文件)。值得一提的是,在注释上,ChIPseeker没有物种限制,当然物种得有那两个文件。
查看peaks在基因组上的分布:
##指定tssRegion(启动子区域),一般TSS上下游区域作为启动子区域
> peakAnno
查看peaks左右5kb区域都有哪些基因:
##addFlankGeneInfo看peak附近基因,flankDistance看peak左右5kb距离
在输出结果中可以看到多出三列,分别是:flank_txIds, flank_geneIds和flank_gene_distances,可以看到在peak附近5kb区域的所有基因。
虽然找到了peak附近基因,但是基因ID我们不认识,这里用annoDb参数进行转换,利用的是TxDb对象,TxDb对象中的基因ID是哪种,annoDb参数就会将基因ID转换为对应的ID。
peakAnno = annotatePeak(f, tssRegion=c(-1000, 1000), TxDb=txdb, annoDb = 'org.Hs.eg.db')
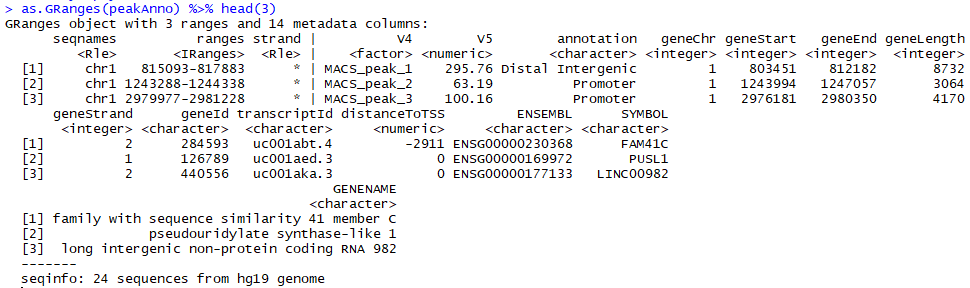
这里,TxDb的基因ID是Entrez,annoDb参数就会将基因ID转成ENSEMBL ID,并且加上对应的注释信息。

0 5
注释信息可视化
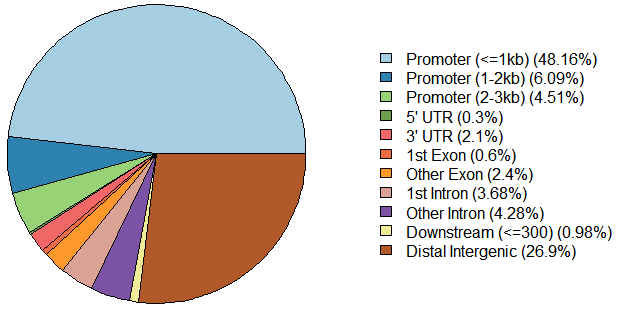
用plotAnnoPie函数可视化peak分布(ceas也可以看):
peakAnno <- annotatePeak(files[[4]], tssRegion =c(-3000, 3000),TxDb=txdb, annoDb="org.Hs.eg.db")
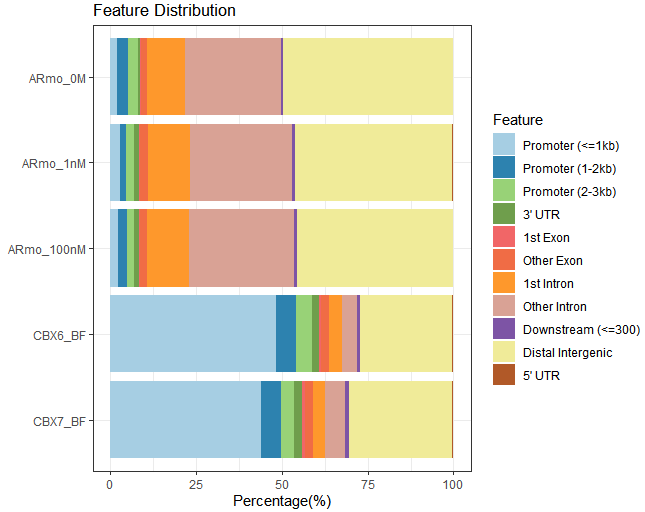
用plotAnnoBar函数同时查看多个样本的peak分布:
peakAnnoList <- lapply(files, annotatePeak,
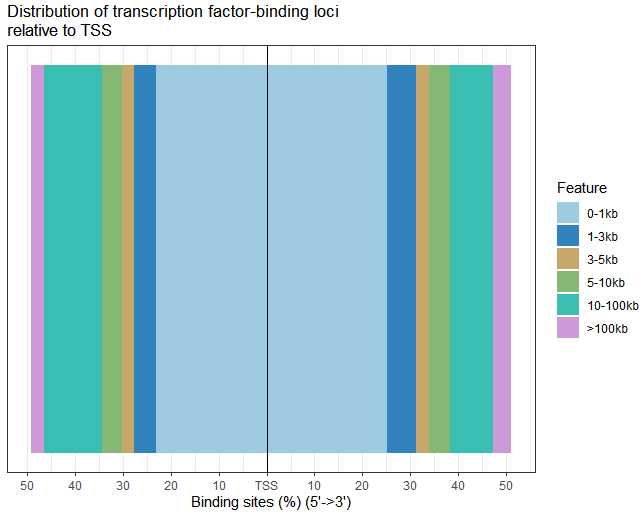
用plotDistToTSS函数可视化peak离最近基因距离的分布:
plotDistToTSS(peakAnno,
用plotDistToTSS函数同时看多个样本peak最近基因距离的分布:
plotDistToTSS(peakAnnoList)
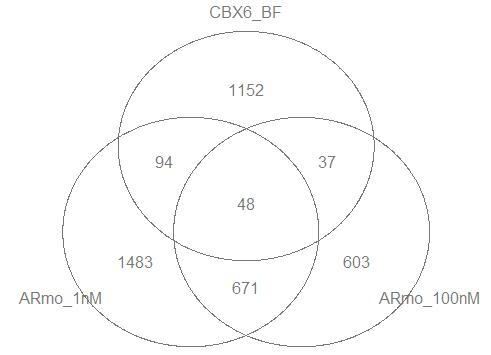
vennplot函数可视化展示注释最近的基因在不同样本中的overlap情况:
genes <- lapply(peakAnnoList, function(i)
//
今天的分享就到这里,有问题请留言吧!
//
我就知道你“在看”

▼更多精彩推荐,请关注我们▼

把时间交给阅读

本文分享自微信公众号 - 生物信息学(swxxx1)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。