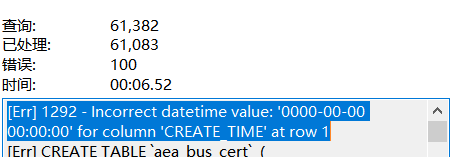
本机系统:Windows 10 (我真是坚持走在用Windows写python的路上不回头。。。)
使用软件:putty(ssh连接) 和 WinSCP(文件管理)
1.登陆
putty软件输入IP地址和端口号, 登陆gwork
2.环境
/ghome下的有一userid命名的文件夹,账号创建即生成,限额20G, 代码及script文件放在这里
/gdata下可以自从创建userid命名的文件夹,限额300G,用于储存job生成的结果。
/gpubg公共数据区
3.使用
调试:从gwork主机sshG101,断开ssh连接的方法,logout
startdocker -u "-it" -c /bin/bash bit:5000/deepo
bit:5000/deepo是一个集中了几乎所有深度学习框架的一个docker 镜像,使用上述命令将进入docker container内部,相当于一个虚拟机,
你在内部对系统所做的任何操作都将在系统退出后丢失,但对用户根目录下(/ghome/<username>)的文件操作将不会丢失。
此命令没有用-P命令挂载用户代码和配置目录,系统默认挂载了整个用户根目录,也没有使用-D挂载数据目录,用户可以根据需要挂载。
退出docker的命令:
exit
退出G101后,查看镜像的命令
sudo docker images
如果没有正确得退出docker(如直接关闭terminal),需要利用命令查看docker id,并且关闭
sudo docker pssudo docker stop CONTAINER_ID
调试成功后,编写pbs文件提交,注意windows系统下的换行符需要改成unix模式(可以使用notepad++编辑器)。
pbs中的命令行如下:
startdocker -D <mydatapath> -P <my-proc-config-path> -s <my-script-file> bit:5000/deepo
""" -s 参数后的my-script-file 可以是shell脚本或python脚本,但都需要在第一行加解释器,如
shell脚本需要加:
#!/bin/bash
python脚本需要加:
#!/usr/local/bin/bash"""
下面是一个pbs文件示例:
#PBS -N myjob1
#PBS -o /home/liulm/myjob1.out
#PBS -e /home/liulm/myjob1.err
#PBS -l nodes=1:gpus=1:S
#PBS -r y
cd $PBS_O_WORKDIR
echo Time is 'date'
echo Directory is $PWD
echo This job runs on the following nodes:
cat $PBS_NODELIFE
cat $PBS_GPULIFE
./myproc"""
-N: job 名
-o: 输出文件路径
-e: 报错文件输出路径
-l: 集群资源分配, 节点数和gpu数(通常一就可以满足要求)
-r: 任务操作类型, -r表示job立即执行
"""
对job的操作指令集合:
qsub myjob1.pbs
#提交作业
qstat -n
#查看作业
qhold job_id
#挂起作业,被挂起的job状态显示为H
qrls job_id
#取消挂起作业,job重新运行
pbsnodes
#查看节点
pbsnodes -l free
#查看空闲节点
4.自定义docker镜像文件(相当于安装库?)
emmm,步骤比较复杂。
而自己python程序中比较小的packeage可以使用如下方法:
不需要构建镜像。以numpy为例:
1. 下载numpy的源码(一般能从个人PC上拷贝),放入服务器上的某个目录,比如/gdata/xxx/pylib
2. 挂载目录,-v /gdata/xxx/pylib:/data/pylib
3. 将挂载后的目录加入python import的搜索路径,两个方法
a. 指定容器的PYTHONPATH, 在startdocker的-u里面加入-e PYTHONPATH=/data/pylib,此时/data/pylib的
优先级可能次于系统自带路径(没测过),import numpy 会导入系统的numpy
b. 在程序的一开始加入下面两行代码
import sys
sys.path.insert(1, '/data/pylib')
1表示/data/pylib的优先级仅次于程序所在目录
此时/data/pylib的优先级高于系统自带路径,import numpy 会导入/data/pylib的numpy
5. DGX1服务器使用
slurm构成的小集群, 操作仍在gwork 上,共享文件系统
script格式如下:
my.slurm
#!/bin/bash
#SBATCH --job-name=myjob1
#SBATCH -gres=gpu:4
#SBATCH --output=myjob.out
echo "Submitted from $SLURM_SUBMIT_DIR" on node "$SLURM_SUBMIT_HOST"
echo "Running on node "$SLURM _JOB_NODELIST
echo "Allocate Gpu Units:" $CUDA_VISABLE_DEVICES
##program here##
startdocker {-c <mycd>|-s <myscript>} nvdl.githost.io:4678/dgx/cuda:9.0-cudnn7-devel-ubuntu16.04
操作命令:
sbatch <myscript>
#提交文件
squeue
#查看队列情况
scancel <jobid>
#取消job